Quantification numérique multiplexée de l’expression des gènes et de la variation du nombre de copies dans les échantillons cliniques
Numérique et automatisé,
reproductible et précis
La technologie NanoStringMC est utilisée pour capturer et dénombrer des molécules d’ADN (CNV, SNV, in/del), d’ARN (ARNm, miARN, fusions) ou des protéines (y compris celles qui présentent des modifications post-traductionnelles) à l’aide d’un système de sondes chromocodées. Les avantages de la technologie NanoStringMC par rapport aux plateformes existantes sont notamment sa capacité à mesurer directement les cibles, sans réactions enzymatiques ni biais d’amplification, sa sensibilité couplée à une haute capacité de multiplexage, et sa production de résultats numériques.
Le système d’analyse de l’expression des gènes nCounter® de NanoStringMC est plus sensible que celui des biopuces et d’une sensibilité et d’une précision semblables à celles de la PCR en temps réel. Comme il permet la quantification des ARNm de faible abondance dans de petits échantillons, ce système est particulièrement utile pour définir des profils d’expression et des signatures spécifiques dans des échantillons cliniques.
CellCarta offre une analyse sensible et reproductible de l’expression génique permettant de tester jusqu’à 800 gènes en une seule réaction à l’aide de la technologie NanoStringMC.
Un code unique est attribué à chaque gène
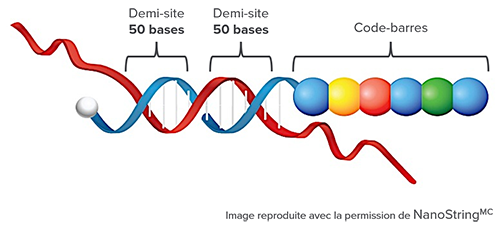
Le système NanoStringMC permet le dénombrement numérisé jusqu’à environ 1 million de molécules par zone d’échantillonnage au moyen de deux sondes spécifiques de la cible. Une première sonde (environ 50 bases) complémentaire à la séquence cible et couplée à la biotine permet de capturer la molécule cible, et une deuxième sonde (environ 50 bases) également complémentaire à la séquence cible, mais couplée à une extension chromocodée, procure le signal de détection. Cette extension chromocodée consiste en une série de segments d’ARN complémentaires transcrits in vitro, chacun marqué par un fluorophore spécifique. L’ordre de présentation crée un code unique qui est utilisé pour identifier la cible.
Une fois la cible liée par les deux sondes, elle est isolée sur une surface recouverte de streptavidine, immobilisée dans un état allongé à l’aide d’un champ électrique et visualisée. L’ordre spécifique des signaux fluorescents permet l’identification et la quantification de la cible.
Multiplexage des échantillons et des cibles
Le système NanostringMC possède une capacité de multiplexage élevée et permet de tester jusqu’à 800 gènes cibles en un seul essai à l’aide du système nCounter®.
Idéal pour définir des signatures d’expression génique dans les échantillons cliniques, 100 ng d’ARN total (dans un volume de 5 à 10 μl) sont généralement nécessaires pour la plupart des panels d’expression génique. Le nombre requis d’échantillons FFIP sur lames de verre dépend du type de biopsie et de la cellularité. Les recommandations suivantes peuvent généralement être utilisées comme point de départ :
- Pour les échantillons réséqués volumineux présentant une cellularité élevée, de 2 à 4 coupes de 5 µm
- Pour les petites biopsies ou les échantillons présentant une faible cellularité, de 4 à 10 coupes de 5 µm
Ce protocole simple permet d’obtenir des résultats sensibles, précis et quantitatifs. Les données numériques obtenues comprennent une liste des gènes examinés et le nombre de fois qu’ils ont été détectés dans les échantillons. Différentes représentations des résultats de l’analyse sont offertes pour faciliter l’interprétation des données, notamment des boîtes à moustache pour chaque groupe de traitement, des diagrammes de dispersion de tous les gènes, des analyses avancées spécifiques, des regroupements de gènes et des cartes de densité.
Panels disponibles
Des panels prêts à l’emploi sont offerts et peuvent être personnalisés en fonction des besoins. Le panel « Pan cancer 770-plex » est un panel particulièrement utilisé dans le cadre d’essais cliniques portant sur l’immunothérapie pour étudier les principales voies de signalisation associées à l’apparition et au développement du cancer.
- Panels d’expression génique nCounter®
- PanCancer IO 360MC: panel d’expression génique de 750 gènes liés au cancer
- PanCancer Immune Profiling : panel de 770 gènes pour profilage immunitaire
- PanCancer Progression : panel de 770 gènes liés à la progression du cancer
- PanCancer Pathway : panel de 770 gènes représentant les voies de signalisation essentielles du cancer
- Test pour le sous-typage des lymphomes et l’identification de la cellule d’origine mis au point pour le système nCounter®
- Test PAM50 pour le sous-typage du cancer du sein