Raccourcissez le cycle de développement de vos médicaments en identifiant et en caractérisant les néo-épitopes directement à partir de tissus à l’aide de la spectrométrie de masse
Découverte de néo-épitopes directement à partir de tissus
POURQUOI PRÉDIRE QUAND ON PEUT OBSERVER ?
La course à l’identification des néo-épitopes est cruciale pour la recherche en immunologie et critique dans le développement de médicaments et de vaccins. Deux approches ont été utilisées pour identifier les néo-épitopes. Au cours des dernières années, de nombreuses sociétés pharmaceutiques et biotechnologiques innovantes ont commencé à reconnaître le pouvoir de la spectrométrie de masse pour identifier les peptides présentés qui déclenchent les réponses des lymphocytes T. Des algorithmes et des modèles de prédiction de séquence ont également été utilisés pour identifier des néo-épitopes, mais ces approches conduisent souvent à des taux élevés de candidats qui ne sont pas nécessairement observés dans les tissus.
Avec NEOEPITOPE DIRECTMC, l’identification directe des néo-épitopes dans les tissus peut être réalisée en utilisant la spectrométrie de masse. Cette approche conduit à des peptides naturellement présentés par opposition à des peptides prédits.
Un cycle de développement plus court et plus fiable
UN CHEMIN VERS DES DÉCOUVERTES QUI EST EFFICACE, PLUS RAPIDE ET PLUS FIABLE.
NEOEPITOPE DIRECTMC a été développé grâce à des années d’expertise en spectrométrie de masse. Les peptides naturellement traités et isolés de tissus sont identifiés en utilisant la chromatographie liquide couplée à la spectrométrie de masse en tandem (CL-SM/SM). Notre processus complet de découverte, qualification et caractérisation des néo-épitopes inclut notamment une analyse comparative avec des tissus non tumoraux adjacents appariés, provenant de notre base de données exclusive de tissus sains : AntigenAtlasMC.
L’AntigenAtlasMC, propriété exclusive de CellCarta, est une base de données exhaustive, à jour et créée à partir de tissus sains qui permet une meilleure interprétation et priorisation des données pour la découverte de néo-épitopes.
Le mode opératoire optimisé de NEOEPITOPE DIRECTMC réduit le matériel et le temps nécessaires à l’identification des néo-épitopes. C’est une solution rigoureuse et reproductible qui offre une voie fiable vers des découvertes en immuno-oncologie. Notre équipe de scientifiques de premier plan a conçu ce mode opératoire pour éliminer la dépendance aux algorithmes de prédictions et obtenir une plus grande variété de néo-épitopes.
Néo-épitopes pertinents dérivés de tissus
UNE PLUS GRANDE SENSIBILITÉ. UNE MEILLEURE PRÉCISION DE MASSE. DE LA BIOINFORMATIQUE AVANCÉE.
Le mode opératoire optimal qui raccourcit le cycle de développement de vos médicaments:
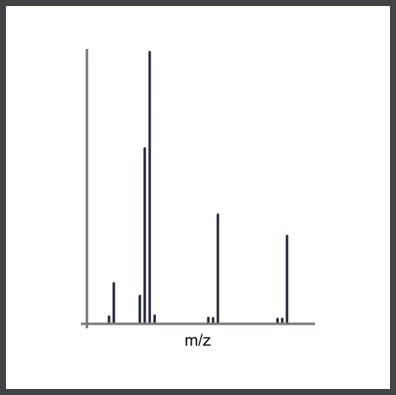
La spectrométrie de masse
(CL-SM/SM) est précise et fiable:
Sensibilité accrue et identification fiable des peptides grâce à des données SM/SM de haute résolution.
AntigenAtlasMC est exhaustif
et à jour: Notre base de données exclusive de tissus sains permet une meilleure interprétation et une meilleure priorisation des données.
L’ELISpot est rapide et efficace : Permet de caractérise la réactivité immunitaire de cibles candidates dans les systèmes autologues ou hétérologues.
Avantages
- Identifie directement les antigènes tumoraux naturellement présentés grâce à une comparaison avec les tissus normaux adjacents
- Se concentre sur les peptides naturellement traités et présentés par des complexes CMH in vivo
- Réduit la dépendance à la modélisation des épitopes
- Offre des candidats mieux définis pour le suivi
- Offre une plus grande couverture de différents types de HLA
Principales applications
- Vaccins thérapeutiques contre le cancer
- Thérapies adoptives fondées sur les lymphocytes-T
- Validation de cibles
Témoignages
“A robust platform that can be depended on to consistently provide high quality data run after run.”
“Peptides identified needed to meet specific length and predict affinity. Meeting these criteria indicated that the peptides identified were high quality, and could be confidently used in further analysis.”